- Plateforme
Epitranscriptomique & Séquençage (EpiRNA-Seq)
- Epitranscriptome, ARN, Séquençage à haut-débit, Modifications post-transcriptionnelles

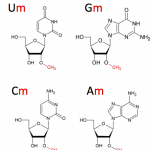
La plateforme Epitranscriptomique et Séquençage (EpiRNA-Seq) est spécialisée et mondialement reconnue dans l'identification et la quantification des nucléotides modifiés au sein des ARN (Epitranscriptomique). Elle s'appuie sur les compétences de l'UMR7365 IMoPA dans le domaine de la biologie moléculaire des ARN.
Installée sur le campus Brabois-Santé de l'Université de Lorraine à Nancy, elle met à disposition de la communauté scientifique académique et industrielle des ressources de haute-technologie (séquenceurs à haut-débit de type Illumina). Depuis 2019, elle est labellisée au niveau local en tant que Structure d'appui à la Recherche (StAR-LUE) dans le cadre du programme INFRA+ de Lorraine Université d'Excellence, et depuis 2021, elle est reconnue au niveau national par le label IBiSA. Elle fait partie du réseau européen COST EPITRAN et organise régulièrement des actions de formation à destination des étudiants et des chercheurs.
La plateforme possède un véritable savoir-faire technique et méthodologique reconnu permettant de contribuer à différents projets d'Epitranscriptomique (RiboMeth-Seq, AlkAniline-Seq, HydraPsi-Seq, RT-Seq) pour aborder des questions scientifiques diverses telles que la dynamique des modifications post-transcriptionnelles dans les ARN.
La plateforme fonctionne principalement en mode collaboratif (implication de la plateforme dans les étapes de votre projet de séquençage de la conception du design expérimental jusqu'à l'analyse statistique des données) mais le mode prestation (principalement séquençage de librairies préparées par vos soins sans implication de la plateforme) est également envisageable dans certaines conditions. La plateforme dispose d'une tarification différentielle (Université de Lorraine, académique et industriel).
La plateforme est ouverte à la communauté scientifique académique et industrielle au niveau local, national et international.
L'équipe est à votre disposition pour vous aider en apportant ses savoir-faires techniques et méthodologiques et vous accompagner de A à Z dans vos projets de séquençage et plus particulièrement d'épitranscriptomique.
La plateforme dispose d'une tarification différentielle (Université de Lorraine, partenaires académiques et industriels).
Tout nouveau collaborateur doit remplir le formulaire de "Demande de prestation" afin de définir la meilleure stratégie à adopter pour la conduite de l’étude, et obtenir un devis. Tout utilisateur s’engagera à accepter les conditions générales (charte utilisateurs). Les instructions pour préparer vos échantillons et les envoyer à la plateforme sont données dans le formulaire "Echantillons".
- Kopietz K, Raorane K, Guo W, Flegler F, Bourguignon V, Thuillier Q, Kilz LM, Weber M, Marchand V, Reuter K, Tuorto F, Helm M, Motorin Y. TGT Damages its Substrate tRNAs by the Formation of Abasic Sites in the Anticodon Loop. J Mol Biol. 2025 Feb 25:169000.
 10.1016/j.jmb.2025.169000 ,
10.1016/j.jmb.2025.169000 ,  40011082 ,
40011082 ,  HAL-05105939
HAL-05105939 - Fruchard L, Babosan A, Carvalho A, Lang M, Li B, Duchateau M, Giai Gianetto Q, Matondo M, Bonhomme F, Hatin I, Arbes H, Fabret C, Corler E, Sanchez G, Marchand V, Motorin Y, Namy O, de Crécy-Lagard V, Mazel D, Baharoglu Z. Aminoglycoside tolerance in Vibrio cholerae engages translational reprogramming associated with queuosine tRNA modification. Elife. 2025 Jan 6;13:RP96317.
 10.7554/eLife.96317 ,
10.7554/eLife.96317 ,  39761105 ,
39761105 ,  HAL-05029047
HAL-05029047 - Kilz LM, Zimmermann S, Marchand V, Bourguignon V, Sudol C, Brégeon D, Hamdane D, Motorin Y, Helm M. Differential redox sensitivity of tRNA dihydrouridylation. Nucleic Acids Res. 2024 Nov 27;52(21):12784-12797.
 10.1093/nar/gkae964 ,
10.1093/nar/gkae964 ,  39460624 ,
39460624 ,  HAL-04911631
HAL-04911631 - Henkel M, Fillbrunn A, Marchand V, Raghunathan G, Berthold MR, Motorin Y, Marx A. A DNA Polymerase Variant Senses the Epigenetic Marker 5-Methylcytosine by Increased Misincorporation. Angew Chem Int Ed Engl. 2024 Nov 25;63(48):e202413304.
 10.1002/anie.202413304 ,
10.1002/anie.202413304 ,  39449390 ,
39449390 ,  HAL-04939883
HAL-04939883 - Dominique C, Maiga NK, Méndez-Godoy A, Pillet B, Hamze H, Léger-Silvestre I, Henry Y, Marchand V, Gomes Neto V, Dez C, Motorin Y, Kressler D, Gadal O, Henras AK, Albert B. The dual life of disordered lysine-rich domains of snoRNPs in rRNA modification and nucleolar compaction. Nat Commun. 2024 Oct 31;15(1):9415.
 10.1038/s41467-024-53805-1 ,
10.1038/s41467-024-53805-1 ,  39482307 ,
39482307 ,  HAL-04797120
HAL-04797120 - Schartel L, Jann C, Wierczeiko A, Butto T, Mündnich S, Marchand V, Motorin Y, Helm M, Gerber S, Lemke EA. Selective RNA pseudouridinylation in situ by circular gRNAs in designer organelles. Nat Commun. 2024 Oct 24;15(1):9177.
 10.1038/s41467-024-53403-1 ,
10.1038/s41467-024-53403-1 ,  39448590 ,
39448590 ,  HAL-04939840
HAL-04939840 - Kristen M, Lander M, Kilz LM, Gleue L, Jörg M, Bregeon D, Hamdane D, Marchand V, Motorin Y, Friedland K, Helm M. DORQ-seq: high-throughput quantification of femtomol tRNA pools by combination of cDNA hybridization and Deep sequencing. Nucleic Acids Res. 2024 Oct 14;52(18):e89.
 10.1093/nar/gkae765 ,
10.1093/nar/gkae765 ,  39258547
39258547 - Rahman ML, Bonnard AA, Wang F, Ruaud L, Guimiot F, Li Y, Defer I, Wang Y, Marchand V, Motorin Y, Yao B, Drunat S, Ghalei H. New ZNHIT3 Variants Disrupting snoRNP Assembly Cause Prenatal PEHO Syndrome with Isolated Hydrops. medRxiv [Preprint]. 2024 Aug 26:2024.08.26.24312490.
 10.1101/2024.08.26.24312490 ,
10.1101/2024.08.26.24312490 ,  39252897
39252897 - Toubdji S, Thullier Q, Kilz LM, Marchand V, Yuan Y, Sudol C, Goyenvalle C, Jean-Jean O, Rose S, Douthwaite S, Hardy L, Baharoglu Z, de Crécy-Lagard V, Helm M, Motorin Y, Hamdane D, Brégeon D. Exploring a unique class of flavoenzymes: Identification and biochemical characterization of ribosomal RNA dihydrouridine synthase. Proc Natl Acad Sci U S A. 2024 Aug 6;121(32):e2401981121.
 10.1073/pnas.2401981121 ,
10.1073/pnas.2401981121 ,  39078675 ,
39078675 ,  HAL-04734945
HAL-04734945 - Sudol C, Kilz LM, Marchand V, Thullier Q, Guérineau V, Goyenvalle C, Faivre B, Toubdji S, Lombard M, Jean-Jean O, de Crécy-Lagard V, Helm M, Motorin Y, Brégeon D, Hamdane D. Functional redundancy in tRNA dihydrouridylation. Nucleic Acids Res. 2024 Jun 10;52(10):5880-5894.
 10.1093/nar/gkae325 ,
10.1093/nar/gkae325 ,  38682613 ,
38682613 ,  HAL-04614743
HAL-04614743

Formation CNRS Entreprises
( -> )
La plateforme EpiRNA-Seq vous propose la formation CNRS Entreprises suivante : "Epitranscriptomics: mapping and analysis of RNA modifications by Next-Generation Sequencing".
Cette formation aura lieu du 17 au 19 mai 2021 sur la plateforme de l'UMS2008/US40 IBSLor au Biopôle à Nancy.
Pour vous inscrire : https://cnrsformation.cnrs.fr/epitranscriptomics-mapping-and-analysis-of-rna-modifications-by-next-generation-sequencing?axe=140

Formation Epitranscriptomique (COST Epitran)
( -> )
La plateforme EpiRNA-Seq organise en collaboration avec I. Motorine (UMR7365 IMoPA) une école thématique Epitranscriptomique avec le soutien du COST Epitran du 10-13 février 2020 à Nancy (France). Cette formation est destinée principalement aux étudiants en thèse et postdocs.