- Plateforme
Epitranscriptomique & Séquençage (EpiRNA-Seq)
- Epitranscriptome, ARN, Séquençage à haut-débit, Modifications post-transcriptionnelles

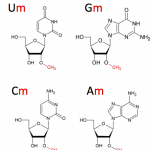
La plateforme Epitranscriptomique et Séquençage (EpiRNA-Seq) est spécialisée et mondialement reconnue dans l'identification et la quantification des nucléotides modifiés au sein des ARN (Epitranscriptomique). Elle s'appuie sur les compétences de l'UMR7365 IMoPA dans le domaine de la biologie moléculaire des ARN.
Installée sur le campus Brabois-Santé de l'Université de Lorraine à Nancy, elle met à disposition de la communauté scientifique académique et industrielle des ressources de haute-technologie (séquenceurs à haut-débit de type Illumina). Depuis 2019, elle est labellisée au niveau local en tant que Structure d'appui à la Recherche (StAR-LUE) dans le cadre du programme INFRA+ de Lorraine Université d'Excellence, et depuis 2021, elle est reconnue au niveau national par le label IBiSA. Elle fait partie du réseau européen COST EPITRAN et organise régulièrement des actions de formation à destination des étudiants et des chercheurs.
La plateforme possède un véritable savoir-faire technique et méthodologique reconnu permettant de contribuer à différents projets d'Epitranscriptomique (RiboMeth-Seq, AlkAniline-Seq, HydraPsi-Seq, RT-Seq) pour aborder des questions scientifiques diverses telles que la dynamique des modifications post-transcriptionnelles dans les ARN.
La plateforme fonctionne principalement en mode collaboratif (implication de la plateforme dans les étapes de votre projet de séquençage de la conception du design expérimental jusqu'à l'analyse statistique des données) mais le mode prestation (principalement séquençage de librairies préparées par vos soins sans implication de la plateforme) est également envisageable dans certaines conditions. La plateforme dispose d'une tarification différentielle (Université de Lorraine, académique et industriel).
La plateforme est ouverte à la communauté scientifique académique et industrielle au niveau local, national et international.
L'équipe est à votre disposition pour vous aider en apportant ses savoir-faires techniques et méthodologiques et vous accompagner de A à Z dans vos projets de séquençage et plus particulièrement d'épitranscriptomique.
La plateforme dispose d'une tarification différentielle (Université de Lorraine, partenaires académiques et industriels).
Tout nouveau collaborateur doit remplir le formulaire de "Demande de prestation" afin de définir la meilleure stratégie à adopter pour la conduite de l’étude, et obtenir un devis. Tout utilisateur s’engagera à accepter les conditions générales (charte utilisateurs). Les instructions pour préparer vos échantillons et les envoyer à la plateforme sont données dans le formulaire "Echantillons".
- Van den Akker GGH, Chabronova A, Housmans BAC, van der Vloet L, Surtel DAM, Cremers A, Marchand V, Motorin Y, Caron MMJ, Peffers MJ, Welting TJM. TGF-β2 Induces Ribosome Activity, Alters Ribosome Composition and Inhibits IRES-Mediated Translation in Chondrocytes. Int J Mol Sci. 2024 May 5;25(9):5031.
 10.3390/ijms25095031 ,
10.3390/ijms25095031 ,  38732249 ,
38732249 ,  HAL-04934350
HAL-04934350 - Sklias A, Cruciani S, Marchand V, Spagnuolo M, Lavergne G, Bourguignon V, Brambilla A, Dreos R, Marygold SJ, Novoa EM, Motorin Y, Roignant JY. Comprehensive map of ribosomal 2'-O-methylation and C/D box snoRNAs in Drosophila melanogaster. Nucleic Acids Res. 2024 Apr 12;52(6):2848-2864.
 10.1093/nar/gkae139 ,
10.1093/nar/gkae139 ,  38416577 ,
38416577 ,  HAL-04934064
HAL-04934064 Quaiyum S, Sun J, Marchand V, Sun G, Reed CJ, Motorin Y, Dedon PC, Minnick MF, de Crécy-Lagard V. Mapping the tRNA modification landscape of Bartonella henselae Houston I and Bartonella quintana Toulouse. Front Microbiol. 2024 Mar 13;15:1369018.
 10.3389/fmicb.2024.1369018 ,
10.3389/fmicb.2024.1369018 ,  38544857 ,
38544857 ,  HAL-04934028
HAL-04934028 - Lucas MC, Pryszcz LP, Medina R, Milenkovic I, Camacho N, Marchand V, Motorin Y, Ribas de Pouplana L, Novoa EM. Quantitative analysis of tRNA abundance and modifications by nanopore RNA sequencing. Nat Biotechnol. 2024 Jan;42(1):72-86.
 10.1038/s41587-023-01743-6 ,
10.1038/s41587-023-01743-6 ,  37024678 ,
37024678 ,  HAL-04067699
HAL-04067699 - Chabronova A, van den Akker G, Housmans BAC, Caron MMJ, Cremers A, Surtel DAM, Peffers MJ, van Rhijn LW, Marchand V, Motorin Y, Welting TJM. Depletion of SNORA33 Abolishes ψ of 28S-U4966 and Affects the Ribosome Translational Apparatus. Int J Mol Sci. 2023 Aug 8;24(16):12578.
 10.3390/ijms241612578 ,
10.3390/ijms241612578 ,  37628759 ,
37628759 ,  HAL-04933978
HAL-04933978 García-Vílchez R, Añazco-Guenkova AM, Dietmann S, López J, Morón-Calvente V, D'Ambrosi S, Nombela P, Zamacola K, Mendizabal I, García-Longarte S, Zabala-Letona A, Astobiza I, Fernández S, Paniagua A, Miguel-López B, Marchand V, Alonso-López D, Merkel A, García-Tuñón I, Ugalde-Olano A, Loizaga-Iriarte A, Lacasa-Viscasillas I, Unda M, Azkargorta M, Elortza F, Bárcena L, Gonzalez-Lopez M, Aransay AM, Di Domenico T, Sánchez-Martín MA, De Las Rivas J, Guil S, Motorin Y, Helm M, Pandolfi PP, Carracedo A, Blanco S. METTL1 promotes tumorigenesis through tRNA-derived fragment biogenesis in prostate cancer. Mol Cancer. 2023 Jul 29;22(1):119.
 10.1186/s12943-023-01809-8 ,
10.1186/s12943-023-01809-8 ,  37516825 ,
37516825 ,  hal-04187091
hal-04187091 - Montacié C, Riondet C, Wei L, Darriere T, Weiss A, Pontvianne F, Escande ML, de Bures A, Jobet E, Barbarossa A, Carpentier MC, Aarts MGM, Attina A, Hirtz C, David A, Marchand V, Motorin Y, Curie C, Mari S, Reichheld JP, Sáez-Vásquez J. nicotianamine synthase activity affects nucleolar iron accumulation and impacts rDNA silencing and RNA methylation in Arabidopsis. J Exp Bot. 2023 May 14:erad180.
 10.1093/jxb/erad180 ,
10.1093/jxb/erad180 ,  37179467 ,
37179467 ,  HAL-04097969
HAL-04097969 - Lucas MC, Pryszcz LP, Medina R, Milenkovic I, Camacho N, Marchand V, Motorin Y, Ribas de Pouplana L, Novoa EM. Quantitative analysis of tRNA abundance and modifications by nanopore RNA sequencing. Nat Biotechnol. 2023 Apr 6.
 10.1038/s41587-023-01743-6 ,
10.1038/s41587-023-01743-6 ,  37024678 ,
37024678 ,  HAL-04067699
HAL-04067699 Huber LB, Kaur N, Henkel M, Marchand V, Motorin Y, Ehrenhofer-Murray AE, Marx A. A dual-purpose polymerase engineered for direct sequencing of pseudouridine and queuosine. Nucleic Acids Res. 2023 Mar 27:gkad177.
 10.1093/nar/gkad177 ,
10.1093/nar/gkad177 ,  36971106 ,
36971106 ,  HAL-04067720
HAL-04067720 - Gawade K, Plewka P, Häfner SJ, Lund AH, Marchand V, Motorin Y, Szczesniak MW, Raczynska KD. FUS regulates a subset of snoRNA expression and modulates the level of rRNA modifications. Sci Rep. 2023 Feb 20;13(1):2974.
 10.1038/s41598-023-30068-2 ,
10.1038/s41598-023-30068-2 ,  36806717 ,
36806717 ,  HAL-04015688
HAL-04015688

Formation CNRS Entreprises
( -> )
La plateforme EpiRNA-Seq vous propose la formation CNRS Entreprises suivante : "Epitranscriptomics: mapping and analysis of RNA modifications by Next-Generation Sequencing".
Cette formation aura lieu du 17 au 19 mai 2021 sur la plateforme de l'UMS2008/US40 IBSLor au Biopôle à Nancy.
Pour vous inscrire : https://cnrsformation.cnrs.fr/epitranscriptomics-mapping-and-analysis-of-rna-modifications-by-next-generation-sequencing?axe=140

Formation Epitranscriptomique (COST Epitran)
( -> )
La plateforme EpiRNA-Seq organise en collaboration avec I. Motorine (UMR7365 IMoPA) une école thématique Epitranscriptomique avec le soutien du COST Epitran du 10-13 février 2020 à Nancy (France). Cette formation est destinée principalement aux étudiants en thèse et postdocs.