- Plateforme
Epitranscriptomique & Séquençage (EpiRNA-Seq)
- Epitranscriptome, ARN, Séquençage à haut-débit, Modifications post-transcriptionnelles

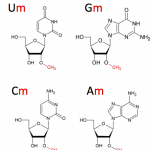
La plateforme Epitranscriptomique et Séquençage (EpiRNA-Seq) est spécialisée et mondialement reconnue dans l'identification et la quantification des nucléotides modifiés au sein des ARN (Epitranscriptomique). Elle s'appuie sur les compétences de l'UMR7365 IMoPA dans le domaine de la biologie moléculaire des ARN.
Installée sur le campus Brabois-Santé de l'Université de Lorraine à Nancy, elle met à disposition de la communauté scientifique académique et industrielle des ressources de haute-technologie (séquenceurs à haut-débit de type Illumina). Depuis 2019, elle est labellisée au niveau local en tant que Structure d'appui à la Recherche (StAR-LUE) dans le cadre du programme INFRA+ de Lorraine Université d'Excellence, et depuis 2021, elle est reconnue au niveau national par le label IBiSA. Elle fait partie du réseau européen COST EPITRAN et organise régulièrement des actions de formation à destination des étudiants et des chercheurs.
La plateforme possède un véritable savoir-faire technique et méthodologique reconnu permettant de contribuer à différents projets d'Epitranscriptomique (RiboMeth-Seq, AlkAniline-Seq, HydraPsi-Seq, RT-Seq) pour aborder des questions scientifiques diverses telles que la dynamique des modifications post-transcriptionnelles dans les ARN.
La plateforme fonctionne principalement en mode collaboratif (implication de la plateforme dans les étapes de votre projet de séquençage de la conception du design expérimental jusqu'à l'analyse statistique des données) mais le mode prestation (principalement séquençage de librairies préparées par vos soins sans implication de la plateforme) est également envisageable dans certaines conditions. La plateforme dispose d'une tarification différentielle (Université de Lorraine, académique et industriel).
La plateforme est ouverte à la communauté scientifique académique et industrielle au niveau local, national et international.
L'équipe est à votre disposition pour vous aider en apportant ses savoir-faires techniques et méthodologiques et vous accompagner de A à Z dans vos projets de séquençage et plus particulièrement d'épitranscriptomique.
La plateforme dispose d'une tarification différentielle (Université de Lorraine, partenaires académiques et industriels).
Tout nouveau collaborateur doit remplir le formulaire de "Demande de prestation" afin de définir la meilleure stratégie à adopter pour la conduite de l’étude, et obtenir un devis. Tout utilisateur s’engagera à accepter les conditions générales (charte utilisateurs). Les instructions pour préparer vos échantillons et les envoyer à la plateforme sont données dans le formulaire "Echantillons".
Pichot F, Marchand V, Helm M, Motorin Y. Data Analysis Pipeline for Detection and Quantification of Pseudouridine (ψ) in RNA by HydraPsiSeq. Methods Mol Biol. 2023;2624:207-223.
 10.1007/978-1-0716-2962-8_14 ,
10.1007/978-1-0716-2962-8_14 ,  36723818 ,
36723818 ,  HAL-03971146
HAL-03971146 - Brazane M, Dimitrova DG, Pigeon J, Paolantoni C, Ye T, Marchand V, Da Silva B, Schaefer E, Angelova MT, Stark Z, Delatycki M, Dudding-Byth T, Gecz J, Plaçais PY, Teysset L, Préat T, Piton A, Hassan BA, Roignant JY, Motorin Y, Carré C. The ribose methylation enzyme FTSJ1 has a conserved role in neuron morphology and learning performance. Life Sci Alliance. 2023 Jan 31;6(4):e202201877.
 10.26508/lsa.202201877 ,
10.26508/lsa.202201877 ,  36720500 ,
36720500 ,  HAL-03967369
HAL-03967369 - Chabronova A, van den Akker GGH, Housmans BAC, Caron MMJ, Cremers A, Surtel DAM, Wichapong K, Peffers MMJ, van Rhijn LW, Marchand V, Motorin Y, Welting TJM. Ribosomal RNA-based epitranscriptomic regulation of chondrocyte translation and proteome in osteoarthritis. Osteoarthritis Cartilage. 2023 Jan 5:S1063-4584(23)00001-8.
 10.1016/j.joca.2022.12.010 ,
10.1016/j.joca.2022.12.010 ,  36621590 ,
36621590 ,  HAL-03930713
HAL-03930713 - Pichot F, Hogg MC, Marchand V, Bourguignon V, Jirström E, Farrell C, Gibriel HA, Prehn JHM, Motorin Y, Helm M. Quantification of substoichiometric modification reveals global tsRNA hypomodification, preferences for angiogenin-mediated tRNA cleavage, and idiosyncratic epitranscriptomes of human neuronal cell-lines. Comput Struct Biotechnol J. 2022 Dec 19;21:401-417.
 10.1016/j.csbj.2022.12.020 ,
10.1016/j.csbj.2022.12.020 ,  36618980 ,
36618980 ,  HAL-03930749
HAL-03930749 Maharana S, Kretschmer S, Hunger S, Yan X, Kuster D, Traikov S, Zillinger T, Gentzel M, Elangovan S, Dasgupta P, Chappidi N, Lucas N, Maser KI, Maatz H, Rapp A, Marchand V, Chang YT, Motorin Y, Hubner N, Hartmann G, Hyman AA, Alberti S, Lee-Kirsch MA. SAMHD1 controls innate immunity by regulating condensation of immunogenic self RNA. Mol Cell. 2022 Sep 16:S1097-2765(22)00851-6.
 10.1016/j.molcel.2022.08.031 ,
10.1016/j.molcel.2022.08.031 ,  36150385 ,
36150385 ,  HAL-03787894
HAL-03787894 Henry BA, Marchand V, Schlegel BT, Helm M, Motorin Y, Lee N. Pseudouridylation of Epstein-Barr Virus Noncoding RNA EBER2 Facilitates Lytic Replication. RNA. 2022 Sep 13:rna.079219.122
 10.1261/rna.079219.122 ,
10.1261/rna.079219.122 ,  36100352 ,
36100352 ,  HAL-03778341
HAL-03778341 Hertler J, Slama K, Schober B, Özrendeci Z, Marchand V, Motorin Y, Helm M. Synthesis of point-modified mRNA. Nucleic Acids Res. 2022 Sep 5:gkac719.
 10.1093/nar/gkac719 ,
10.1093/nar/gkac719 ,  36062567 ,
36062567 ,  HAL-03778358
HAL-03778358 - Dreggors-Walker RE, Cohen LN, Khoshnevis S, Marchand V, Motorin Y, Ghalei H. Studies of mutations of assembly factor Hit 1 in budding yeast suggest translation defects as the molecular basis for PEHO syndrome. J Biol Chem. 2022 Jul 14:102261.
 10.1016/j.jbc.2022.102261 ,
10.1016/j.jbc.2022.102261 ,  35843310 ,
35843310 ,  HAL-03727002
HAL-03727002 - Brégeon D, Pecqueur L, Toubdji S, Sudol C, Lombard M, Fontecave M, de Crécy-Lagard V, Motorin Y, Helm M, Hamdane D. Dihydrouridine in the Transcriptome: New Life for This Ancient RNA Chemical Modification. ACS Chem Biol. 2022 Jun 23.
 10.1021/acschembio.2c00307 ,
10.1021/acschembio.2c00307 ,  35737906 ,
35737906 ,  HAL-03708292
HAL-03708292 Helm M, Motorin Y. Phosphorylation found inside RNA. Nature. 2022 May;605(7909):234-235.
 10.1038/d41586-022-01021-6 ,
10.1038/d41586-022-01021-6 ,  35478019 ,
35478019 ,  HAL-03710903
HAL-03710903

Formation CNRS Entreprises
( -> )
La plateforme EpiRNA-Seq vous propose la formation CNRS Entreprises suivante : "Epitranscriptomics: mapping and analysis of RNA modifications by Next-Generation Sequencing".
Cette formation aura lieu du 17 au 19 mai 2021 sur la plateforme de l'UMS2008/US40 IBSLor au Biopôle à Nancy.
Pour vous inscrire : https://cnrsformation.cnrs.fr/epitranscriptomics-mapping-and-analysis-of-rna-modifications-by-next-generation-sequencing?axe=140

Formation Epitranscriptomique (COST Epitran)
( -> )
La plateforme EpiRNA-Seq organise en collaboration avec I. Motorine (UMR7365 IMoPA) une école thématique Epitranscriptomique avec le soutien du COST Epitran du 10-13 février 2020 à Nancy (France). Cette formation est destinée principalement aux étudiants en thèse et postdocs.