Plateforme
Epitranscriptomique & Séquençage (EpiRNA-Seq)
Epitranscriptomique / Analyse des modifications des ARN
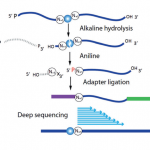
Ces analyses portent principalement sur 3 techniques épitranscriptomiques (RiboMeth-Seq, HydraPsi-Seq et AlkAniline-Seq) développées par la plateforme en étroite collaboration avec l'équipe du Pr Yuri Motorin (UMR7365 IMoPA, CNRS-UL). Pour vous offrir un service de qualité en épitranscriptomique, nous développons ou adaptons d'autres protocoles publiés dans la littérature.
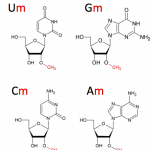
Les technique RiboMeth-Seq et HydraPsi-Seq permettent d'identifier et de quantifier les résidus 2'-O-methylés et les pseudouridines présents dans les ARN respectivement, à partir de seulement 10-50 ng d'ARN totaux ou d'ARN enrichis, tandis que la méthode AlkAniline-Seq permet de localiser les résidus m7G, m3C et D internes dans tous les types d'ARN sans enrichissement préalable à partir de 100-300 ng d'ARN totaux.
- Demande de prestation155.41 Ko
- Charte des utilisateurs667.51 Ko
- Échantillons186.79 Ko
Ces analyses sont réalisées en mode collaboration. Pour plus de renseignements, merci de contacter les responsables de ce service.
Marchand V, Pichot F, Neybecker P, Ayadi L, Bourguignon-Igel V, Wacheul L, Lafontaine DLJ, Pinzano A, Helm M, Motorin Y. HydraPsiSeq: a method for systematic and quantitative mapping of pseudouridines in RNA. Nucleic Acids Res. 2020 Sep 25 : gkaa769.
 10.1093/nar/gkaa769 ,
10.1093/nar/gkaa769 ,  32976574 ,
32976574 ,  HAL-02957799
HAL-02957799 Marchand V, Ayadi L, Ernst FGM, Hertler J, Bourguignon-Igel V, Galvanin A, Kotter A, Helm M, Lafontaine DLJ, Motorin Y. AlkAniline-Seq: profiling of m7G and m3C RNA modifications at single nucleotide resolution. Angew Chem Int Ed Engl.2018 Oct 29.
 10.1002/anie.201810946 ,
10.1002/anie.201810946 ,  30370969 ,
30370969 ,  HAL-01928435
HAL-01928435 Marchand V, Ayadi L, El Hajj A, Blanloeil-Oillo F, Helm M, Motorin Y. High-Throughput Mapping of 2'-O-Me Residues in RNA Using Next-Generation Sequencing (Illumina RiboMethSeq Protocol). Methods Mol Biol. 2017 ; 1562:171-187.
 10.1007/978-1-4939-6807-7_12 ,
10.1007/978-1-4939-6807-7_12 ,  28349461 ,
28349461 ,  HAL-01799289
HAL-01799289 Marchand V, Pichot F, Thüring K, Ayadi L, Freund I, Dalpke A, Helm M, Motorin Y. Next-Generation Sequencing-Based RiboMethSeq Protocol for Analysis of tRNA 2'-O-Methylation. Biomolecules. 2017 Feb 9 ; 7(1).
 10.3390/biom7010013 ,
10.3390/biom7010013 ,  28208788 ,
28208788 ,  HAL-01799272
HAL-01799272 Sharma S, Marchand V, Motorin Y, Lafontaine DLJ. Identification of sites of 2'-O-methylation vulnerability in human ribosomal RNAs by systematic mapping. Sci Rep. 2017 Sep 13 ; 7(1):11490.
 10.1038/s41598-017-09734-9 ,
10.1038/s41598-017-09734-9 ,  28904332 ,
28904332 ,  HAL-01799319
HAL-01799319 Erales J, Marchand V, Panthu B, Gillot S, Belin S, Ghayad SE, Garcia M, Laforêts F, Marcel V, Baudin-Baillieu A, Bertin P, Couté Y, Adrait A, Meyer M, Therizols G, Yusupov M, Namy O, Ohlmann T, Motorin Y, Catez F, Diaz JJ. Evidence for rRNA 2'-O-methylation plasticity: Control of intrinsic translational capabilities of human ribosomes. Proc Natl Acad Sci U S A. 2017 Dec 5 ; 114(49):12934-12939.
 10.1073/pnas.1707674114 ,
10.1073/pnas.1707674114 ,  29158377 ,
29158377 ,  HAL-01866982
HAL-01866982 Ringeard M, Marchand V, Decroly E, Motorin Y, Bennasser Y. FTSJ3 is an RNA 2'-O-methyltransferase recruited by HIV to avoid innate immune sensing. Nature. 2019 Jan 9.
 10.1038/s41586-018-0841-4 ,
10.1038/s41586-018-0841-4 ,  30626973 ,
30626973 ,  HAL-01981983
HAL-01981983 Galvanin A, Ayadi L, Helm M, Motorin Y, Marchand V. Mapping and Quantification of tRNA 2'-O-Methylation by RiboMethSeq. Methods Mol Biol. 2019 ; 1870:273-295.
 10.1007/978-1-4939-8808-2_21 ,
10.1007/978-1-4939-8808-2_21 ,  30539563 ,
30539563 ,  HAL-01957102
HAL-01957102 Ayadi L, Motorin Y, Marchand V. Quantification of 2'-O-Me Residues in RNA Using Next-Generation Sequencing (Illumina RiboMethSeq Protocol). Methods Mol Biol. 2018 ; 1649:29-48.
 10.1007/978-1-4939-7213-5_2 ,
10.1007/978-1-4939-7213-5_2 ,  29130188 ,
29130188 ,  HAL-01661952
HAL-01661952